
Hot
馬爾文帕納科為藥物篩選按下快進鍵!

利用生物物理片段篩選“甜蜜點”
發現抑制BRPF1 的新化學型
馬爾文帕納科為藥物篩選按下快進鍵!
基于化合物庫的有效采樣是片段篩選命中 (fragment screening hit) 識別范式的核心。通常片段分子越小,采樣效率越高。對于小分子和靶蛋白之間弱結合力的檢測能力決定了片段分子可以設計的大小。
CLS*的片段篩選平臺
馬爾文帕納科旗下 Concept Life Sciences 和 Creoptix 聯手開發了一個*的片段篩選平臺。該平臺將專門構建的片段分子庫與光柵耦合干涉 (GCI) 技術相結合,通過在生物物理“甜蜜點” (Sweet Spot) 進行篩選操作,可在幾天內確定有效的片段命中 (fragment hits),從而為片段命中優化提供理想的起點,并為候選藥物提名提供了更快的途徑。
本文報告了使用該平臺進行片段分子篩選,并識別了能有效結合 BRPF1 溴結構域的新片段。
BRPF1
Bromodomains 溴結構域是進化上保守的蛋白質-蛋白質相互作用模塊,可識別組蛋白乙酰化賴氨酸。溴結構域和 PHD 指蛋白 1 (BRPF1) 是單核細胞白血病鋅指 (MOZ) 組蛋白乙酰轉移酶的亞基。它通過多個表觀遺傳閱讀器結構域調節基因轉錄,包括*的雙 PHD 和鋅指組裝、溴結構域和 C 末端 PWWP 結構域(如圖 1所示)。
BRPF1 被認為是肝細胞癌[1]和急性髓細胞白血病[2]的治療靶點,目前這種侵襲性癌癥的成人 5 年生存率不到 30%[3]。
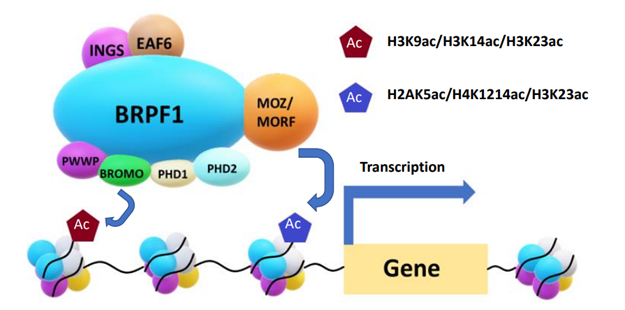
圖 1. MOZ/MORF 組蛋白乙酰轉移酶復合物介導 H3K9、H3K14 和 H3K23 乙酰化以激活基因。BRPF1 包含兩個植物同源域 (PHD) 手指的乙酰閱讀器結構域,它們由一個鋅指節 (PZP 結構域)、一個溴結構域和一個脯氨酸-色氨酸-色氨酸-脯氨酸 (PWWP) 結構域隔開。
近年來報道了一系列 BRPF1 抑制劑,包括 GSK6853[4]和 NI-57 [5](圖 2)。高通量片段對接 [6] 和基于配體的篩選 [7] 也被用于鑒定其他化學型 ,并在 X 射線結構的輔助下對其進行優化(圖 2)。但是目前尚未有選擇性的 BRPF1 抑制劑進入臨床研究,所以能抑制這一靶點的新化學型仍是研究熱點。

圖 2. 左:選擇性 BRPF1 抑制劑的化學結構。右圖:BRPF1與片段分子結合的 X 射線結構 (pdb = 5EQ1)。BRPF1 以綠色帶狀表示,其表面由位于酰基結合域的殘基側鏈構成碳原子為綠色,氧原子為紅色,氮原子為藍色。片段分子以棒狀表示。
生物物理“甜蜜點”
Sweet Spot
據新文獻估計,化學宇宙已經由多達 11 個重原子、1420 萬個化合物增長到了多達 17 個重原子、超過 1660 億個化合物[8]。這種指數增長體現了片段分子大小的增加對化學領域的影響,而片段庫的過分龐雜并不利于基于片段的藥物設計 (FBDD) (圖 3)。伴隨著FBDD的發展,涌現出了多種生物物理技術能夠檢測到片段分子與靶點之間微弱的結合。因此,一個有效的篩選活動應能在篩選化合物時,可以最大限度的在生物物理技術可以檢測到的極限范圍內進行采樣,即片段分子的生物物理“甜蜜點”(圖 3)。

圖 3. 化學宇宙的演變(橙色)、找到篩選命中的機會(灰色)和典型的親和力(藍色)與重原子數量的關系。雖然典型的商業片段庫(紫色)傾向于集合較大的化合物,但通過高靈敏的生物物理技術,在生物物理“甜蜜點”(綠色)范圍內,可以對更小的片段分子進行更有效的篩選。
生物物理片段庫構建

圖 4. 片段庫構建流程:從 55,000 個 CLS 化合物集合到最終 1,133 個選定片段。該過程包括一個生物物理 MPO 對匹配生物物理“甜蜜點”的片段進行分類,以最大限度地提高多樣性,并基于溶解度和純度對化合物質量進行嚴格過濾和篩選。
最da化的化合物多樣性

圖 5.(A)CLS化合物庫的主要成分分析。散點之間間隔越遠,相應片段的結構越多樣化(顏色對應于簇 #),(B)慣性矩陣圖表示 CLS 片段分子的桿狀、片狀和球形形狀(顏色對應于簇 #),(C ) 結構多樣性由在不同 Tanimoto 指數下屬于 1 到 5 個簇的化合物數量表示,(D) 1,133 個 CLS 片段的每個主要chemical handles的百分比。
片段命中類庫
Fragment Hit-Like Library
在過去 20 年的 FBDD 研究中,已報告的片段命中 [9] 和商業片段集合的物理化學特征之間出現了明顯的二分法,這通常由 3 規則 (Ro3) [10] (圖 6) 支持 。
通過瞄準生物物理“甜蜜點”,CLS 生物物理片段集合(圖 6)不僅可以有效地對可用的化合物庫進行采樣,而且還顯示了與報告的片段命中非常匹配的物理化學特征。

圖 6. Giordanetto 等人報道的 486 個片段命中的物理化學特征[9](綠色)、典型的商業片段集合(橙色,從 11 家供應商的 120,000 多個片段中收集)和Concept Life Sciences構建的生物物理片段集合(藍色)。
生物物理篩選

圖 7. SPR 和 GCI 篩選工作流程。GCI 技術使用Creoptix WAVE delta 系統(圖片),該系統可實現簡單且省時的片段篩選。
使用 waveRAPID®
進行動力學篩選

Creoptix WAVE Delta
GCI 分子間相互作用分析儀
這是一種測量動力學的新方法, 不依賴于梯度濃度滴定。waveRAPID®脈沖式注入單一濃度樣品,通過增加樣品在傳感器表面的滯留時間,即可從單個樣品孔中獲得動力學(圖 8)。由于 GCI 的高靈敏度,片段命中可以根據更豐富的數據進行排名,并根據動力學速率常數而不是單獨的親和力來選擇命中。

圖 8. 傳統動力學檢測與 waveRAPID® 比較。在典型的配體結合分析中(左),分析物以增加的濃度加入,每次注入的持續時間一致。在waveRAPID ®分析中(右),該系列由一個循環組成,其中只有一個被解離部分中斷的結合階段和一個最終解離階段。模型擬合用解離階段來完成。
實驗設計和初步篩選數據
基于動力學速率常數擬合、Rmax 和 KD 的命中選擇
使用 2 個傳感器芯片、1100 個移液器吸頭、4 個樣品板和 2.1L 緩沖液,運行時間為 54 小時,可提供完整的動力學信息。


圖 9:waveRAPID® 實驗結果。(A) 在基于GCI技術的 WAVE delta 4 個不同通道上設置的實驗設計。(B) 使用 WAVE control工作站 的 Direct Kinetics 引擎(黑色)將waveRAPID® 傳感圖(紅色)擬合到一對一綁定模型的示例,并提供 Rmax、ka(結合速率)、kd(解離速率)和 KD( 解離平衡常數)。(C) 生成的 waveRAPID® 速率圖,顯示每個片段結合劑的 kd vs ka(顏色為 kd )。產生的 KD 范圍用虛線突出顯示。
參考文獻:
(1) Cheng, C.LH., et. al. Bromodomain-containing protein BRPF1 is a therapeutic target for liver cancer. Commun. Biol. 4, 888 (2021)
(2) Shima, H., et. al. Bromodomain-PHD finger protein 1 is critical for leukemogenesis associated with MOZ-TIF2 fusion. Int J Hematol. 99, 21-31 (2014).
(3) cancerresearchuk.org/about-cancer/acute-myeloid-leukaemia-aml
(4) Bramborough, P., et. al. GSK6853, a Chemical Probe for Inhibition of the BRPF1 Bromodomain. ACS Med. Chem. Lett. 7, 6, 552–557 (2016)
(5) Igoe, N., et. al. Design of a Biased Potent Small Molecule Inhibitor of the Bromodomain and PHD Finger-Containing (BRPF) Proteins Suitable for Cellular and in Vivo Studies. J. Med. Chem. 60, 2, 668–680 (2017)
(6) Zhu, J., et. al. Twenty Crystal Structures of Bromodomain and PHD Finger Containing Protein 1 (BRPF1)/Ligand Complexes Reveal Conserved Binding Motifs and Rare Interactions. J. Med. Chem. 59, 11, 5555–5561 (2016)
(7) Zhu, J., et. al. Structure-based discovery of selective BRPF1 bromodomain inhibitors. Eur. J. Med. Chem. 155, 337-352 (2018)
(8) Ruddigkeit, L., et. al. Enumeration of 166 billion organic small molecules in the chemical universe database GDB-17. J. Chem. Inf. Model., 26, 52, 2864-75 (2012)
(9) Giordanetto, F., et. al. Fragment Hits: What do They Look Like and How do They Bind? J. Med. Chem. 62, 3381−3394 (2019)
(10) Congreve, M., et. al. A 'Rule of Three' for fragment-based lead discovery. Drug Discov. Today 8, 876–877 (2003